SOS反应是细菌应对DNA损伤的整体反应(图1)。LexA蛋白作为高度保守的阻遏蛋白,调控50多个SOS反应相关基因的表达。在正常生长状态下,LexA以二聚体的形式与启动子区的保守DNA序列(SOS box)结合,抑制下游基因的转录。当细菌出现DNA损伤时,RecA蛋白结合ATP和单链DNA(ssDNA),并在DinI的帮助下形成RecA纤维。随后,RecA纤维促进LexA蛋白自我切割来消除对下游SOS反应相关基因的阻遏作用。在SOS反应的后期,RecA纤维还能促进UmuD和λ噬菌体阻遏蛋白CI自我切割,从而诱导细菌的跨损伤修复和噬菌体的裂解周期。鉴于SOS反应可以加速细菌产生耐药突变,其阻断剂的开发日益受到国内外的重视。
2023年1月4日,浙江大学基础医学院冯钰和公共卫生学院周春团队联合攻关,在《PNAS》发表了题为“Structural basis for regulation of SOS response in bacteria”的研究论文,该研究首次解析了细菌SOS反应核心组分RecA蛋白与DinI、LexA、UmuD以及λCI的复合物冷冻电镜结构,统一了细菌SOS反应50年来的研究成果,并为细菌耐药阻断剂的理性设计提供了结构信息。

DinI、LexA、UmuD、λCI结构各异,RecA纤维如何识别这些蛋白?如何促进LexA、UmuD、λCI的自我切割?为了回答这些问题,作者利用螺旋三维重构的方法,分别解析了RecA纤维与DinI、LexA、UmuD、λCI的复合物冷冻电镜结构(图1)。结构显示RecA纤维呈右手螺旋,DinI、LexA、UmuD、λCI则结合在螺旋的沟槽里。
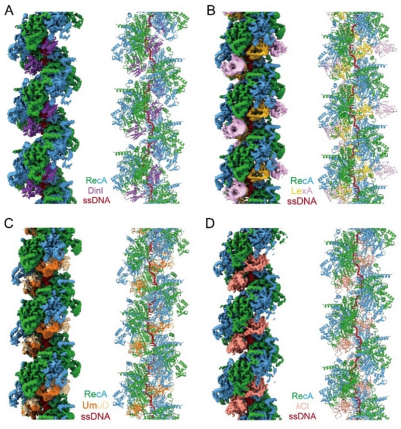
图1 RecA与DinI、LexA、UmuD、λCI的复合物冷冻电镜结构
尽管DinI、LexA、UmuD、λCI的折叠方式和聚集状态各不一样,但是RecA纤维都是通过沟槽里两个高度保守的结构元件识别目标蛋白,其疏水的L2环插入目标蛋白的疏水口袋,其带正电荷的氨基酸簇则与目标蛋白带负电荷的氨基酸簇形成静电相互作用,正是这些疏水和静电作用保证了目标蛋白结合的特异性(图2)。此外,RecA通过范德华力将被切割肽段稳定在易于切割的构象,从而加速LexA、UmuD和λCI的切割。
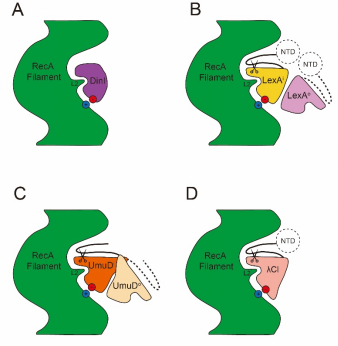
图2 RecA与DinI、LexA、UmuD、λCI相互作用示意图
基因突变是细菌耐药的重要驱动力,而SOS反应又是细菌基因突变的主要来源,因此阻断细菌SOS反应可能减缓细菌耐药的产生,该研究为设计细菌SOS反应的阻断剂提供了精确的结构信息,提示RecA沟槽里的L2环和带正电荷的氨基酸簇可以作为药物开发的靶点。
浙江大学基础医学院博士研究生高波、梁亮和公共卫生学院博士研究生苏璐为该研究的共同第一作者,浙江大学基础医学院冯钰研究员和公共卫生学院周春研究员为该研究的通讯作者。该研究的冷冻电镜工作全部在浙江大学冷冻电镜中心完成,也得到了浙江大学冷冻电镜中心高性能计算平台和医学院蛋白质平台支持。
原文链接:www.pnas.org/doi/10.1073/pnas.2217493120












