2021年2月18日,浙江大学基础医学院、浙江大学医学院附属邵逸夫医院冯钰课题组在Nature Communications在线发表了题为“Transcription activation by a sliding clamp”的研究论文,首次解析了T4噬菌体晚期基因转录激活复合物的冷冻电镜结构,揭示了依赖滑动夹的转录激活机制。
T4噬菌体是分子生物学研究的模式生物之一。T4噬菌体早期基因的转录利用的是细菌的σ因子与RNA聚合酶形成全酶,从而特异性地识别早期基因启动子的-10区和-35区来起始转录,而噬菌体中期和晚期基因启动子没有保守的-35区,所以起始转录的机制有所不同。近年来,冯钰课题组系统地研究了T4噬菌体中期和晚期基因转录激活的分子机制。2019年8月8日,课题组在Nucleic Acids Research发表了题为“Structural basis of σ appropriation”的研究论文,揭示了噬菌体蛋白MotA和AsiA劫持细菌RNA聚合酶激活中期基因转录的分子机制。
2021年2月18日,课题组再次在Nature Communications发文揭示T4噬菌体依赖滑动夹激活晚期基因转录的机制。噬菌体蛋白gp55和细菌RNA聚合酶可以共同起始晚期基因的基础转录,而转录的激活还需要滑动夹gp45(滑动夹是一种环状三聚体蛋白,通常结合在DNA聚合酶上增加其持续合成DNA的能力)以及辅助激活蛋白gp33的参与。课题组利用冷冻电镜单颗粒三维重构的方法,分别解析了T4噬菌体晚期基因的基础转录复合物和转录激活复合物的结构(图1)。
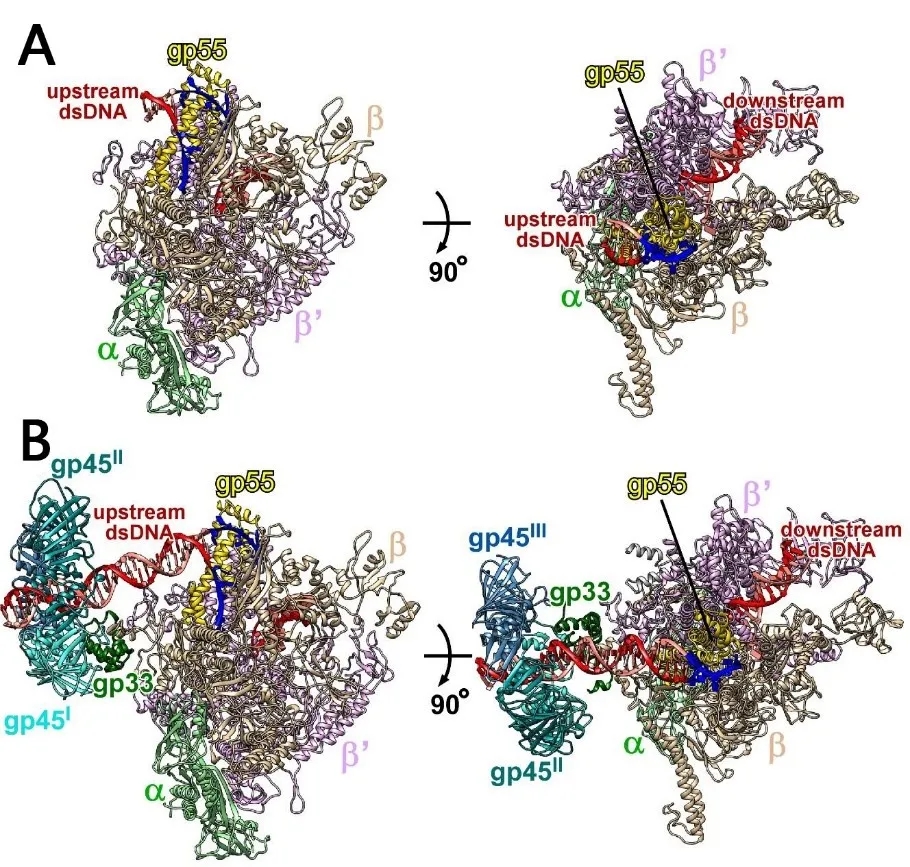
图1 基础转录复合物(A)和转录激活复合物(B)的结构
基础转录复合物的冷冻电镜结构证实了gp55是一个高度分化的σ70家族蛋白,与宿主RNA聚合酶形成全酶,识别并结合晚期基因启动子的-10-like区来起始基础转录。转录激活复合物的冷冻电镜结构显示RNA聚合酶上游的DNA从gp45环中心穿过,辅助激活蛋白gp33则位于gp45环和RNA聚合酶之间。文章在前人的研究基础上,提出了T4噬菌体依赖滑动夹的转录激活模型(图2)。首先,装载器gp44/gp62将gp45装载到DNA上。然后,RNA聚合酶通过gp55和gp33与gp45相连并随着gp45在DNA上滑动,在到达启动子位置后即起始转录。转录延伸过程中gp55与RNA聚合酶脱离,但仍然连接在gp45上。当转录终止后,整个四元复合物不用从DNA上解离即开始下一轮的启动子扫描。滑动夹gp45的存在使得启动子扫描的过程从三维变成了一维,从而大大提高了转录起始的效率。
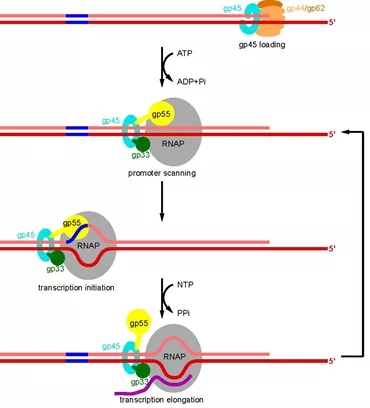
图2 T4噬菌体依赖滑动夹的转录激活模型
浙江大学基础医学院博士后史婧、博士研究生温瑷嘉、硕士研究生金莎为该研究的共同第一作者,浙江大学基础医学院冯钰研究员为该研究的通讯作者。该研究的冷冻电镜工作全部在浙江大学冷冻电镜中心完成,也得到了浙江大学冷冻电镜中心高性能计算平台和医学院蛋白质平台支持。
原文链接:https://doi.org/10.1038/s41467-021-21392-0












